Dna Replikation Bei Escherichia Coli

DNA-Replikation ist ein grundlegender Prozess, der sicherstellt, dass bei der Zellteilung von Escherichia coli (E. coli) jede Tochterzelle eine vollständige und genaue Kopie des genetischen Materials erhält. Da E. coli ein Bakterium ist, also ein Prokaryot, unterscheidet sich die DNA-Replikation in einigen wesentlichen Aspekten von der in eukaryotischen Zellen. Dieser Artikel erklärt den Prozess der DNA-Replikation in E. coli detailliert und verständlich.
Die Grundlagen der DNA-Replikation
Bevor wir uns den spezifischen Mechanismen in E. coli zuwenden, ist es wichtig, die grundlegenden Prinzipien der DNA-Replikation zu verstehen. DNA besteht aus zwei komplementären Strängen, die umeinander gewunden sind und eine Doppelhelix bilden. Jeder Strang besteht aus einer Kette von Nukleotiden, wobei jedes Nukleotid aus einem Desoxyribose-Zucker, einer Phosphatgruppe und einer von vier Stickstoffbasen besteht: Adenin (A), Thymin (T), Cytosin (C) und Guanin (G). A paart sich immer mit T, und C paart sich immer mit G. Diese Basenpaarungsregel ist entscheidend für die DNA-Replikation.
Die DNA-Replikation ist ein semikonservativer Prozess. Das bedeutet, dass jede neue DNA-Doppelhelix einen ursprünglichen (elterlichen) Strang und einen neu synthetisierten Strang enthält. Dies garantiert, dass die genetische Information genau weitergegeben wird.
Initiation der Replikation in E. coli
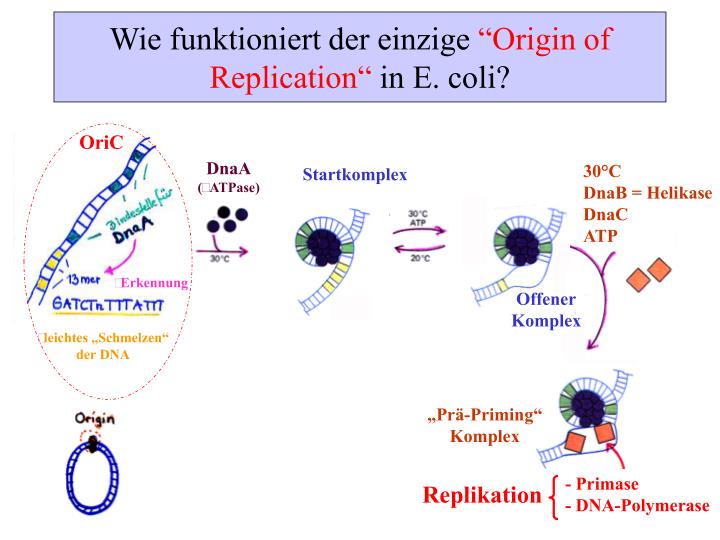
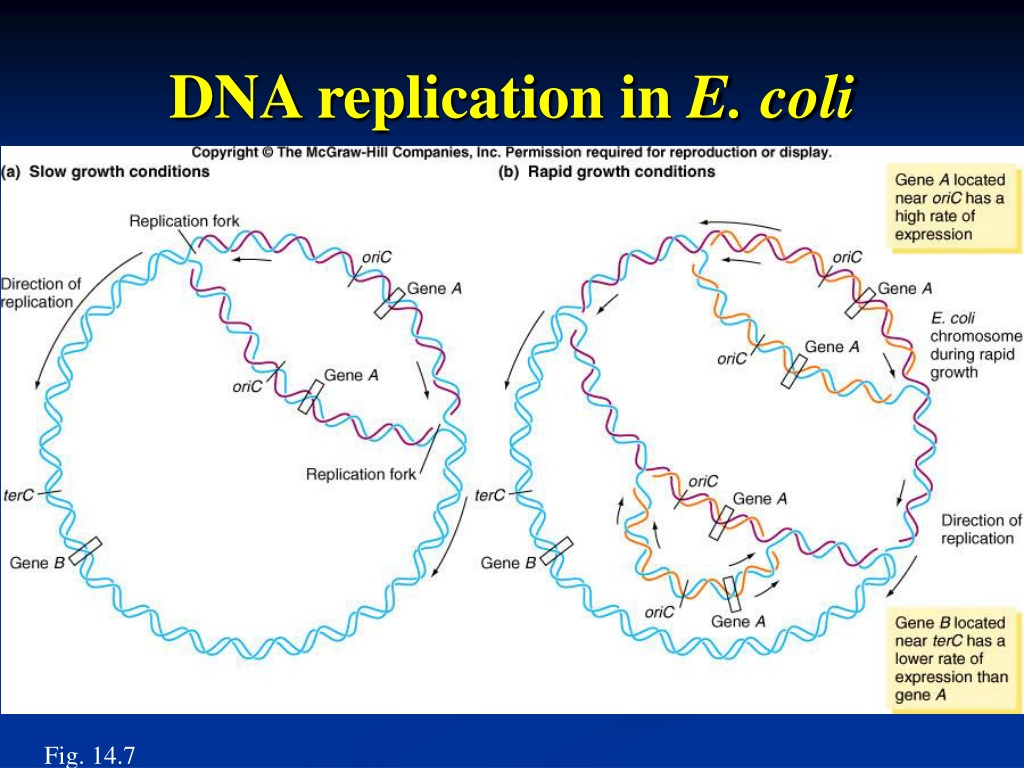
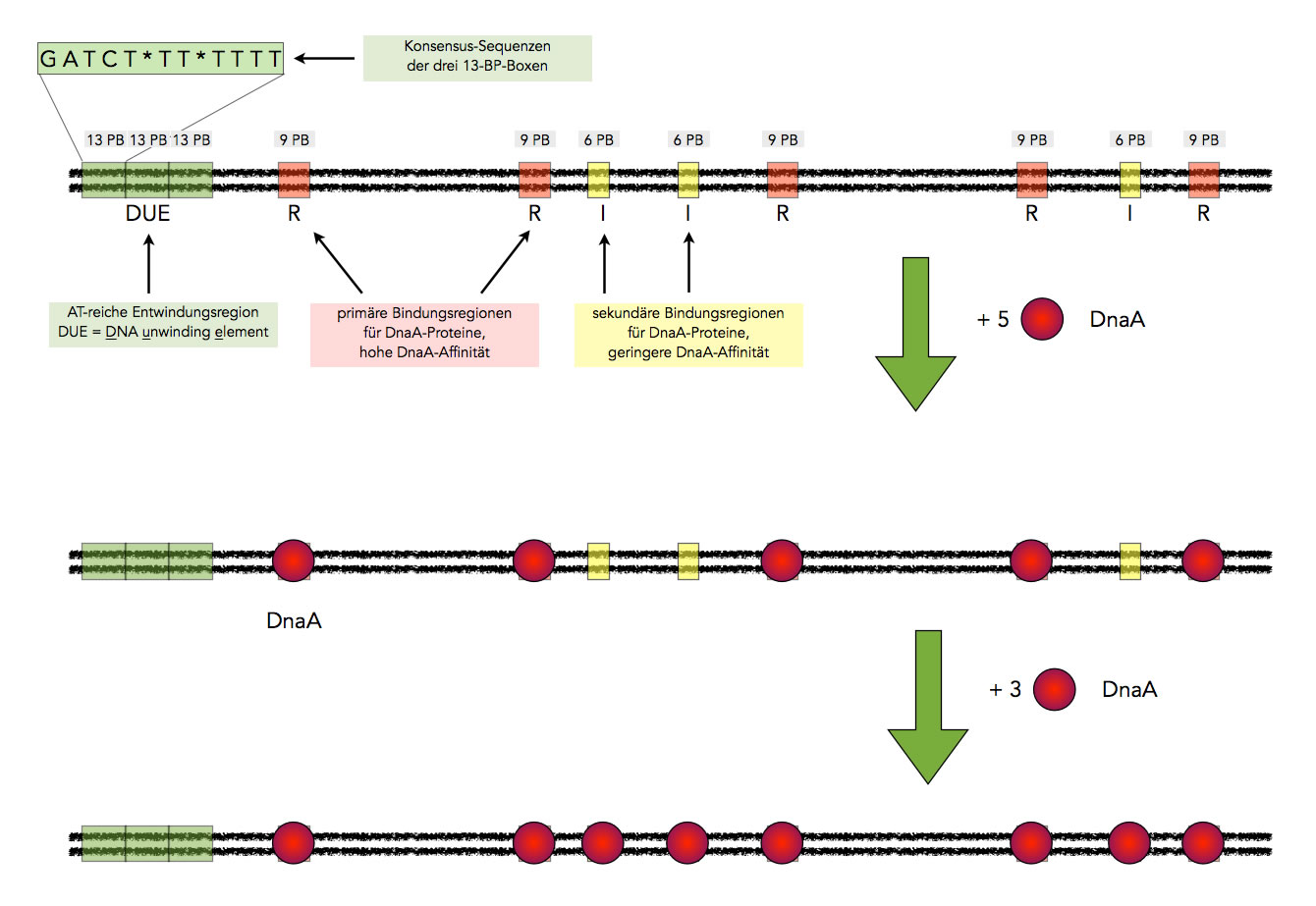
Der Prozess der DNA-Replikation in E. coli beginnt an einem spezifischen Ort auf dem Bakterienchromosom, der als Replikationsursprung (oriC) bezeichnet wird. oriC ist eine etwa 245 Basenpaare lange Sequenz, die reich an A-T-Basenpaaren ist, was die Trennung der DNA-Stränge erleichtert. Die Initiation der Replikation ist ein hochregulierter Prozess, der sicherstellt, dass die DNA nur einmal pro Zellteilung repliziert wird.
Der Prozess läuft wie folgt ab:
- DnaA-Protein-Bindung: Das Initiatorprotein DnaA bindet an spezifische DNA-Sequenzen in oriC. Diese Bindung führt zu einer Konformationsänderung in der DNA, wodurch sich die Doppelhelix an dieser Stelle entwindet.
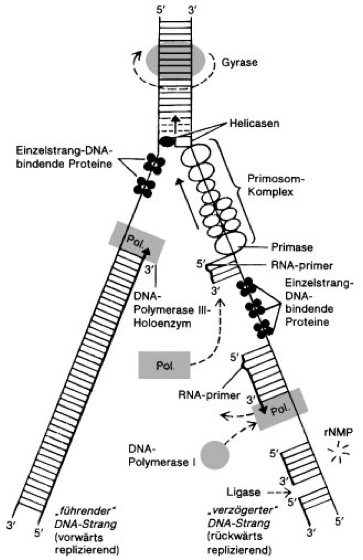
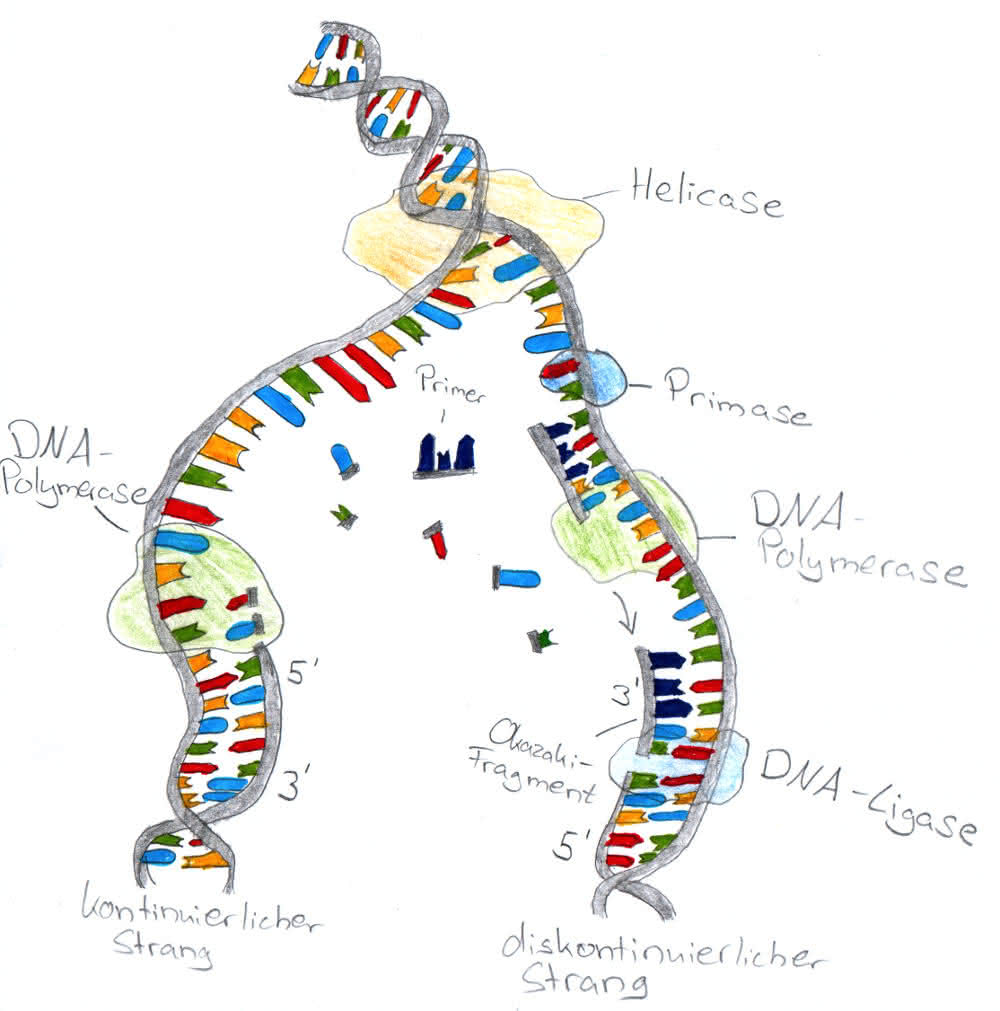
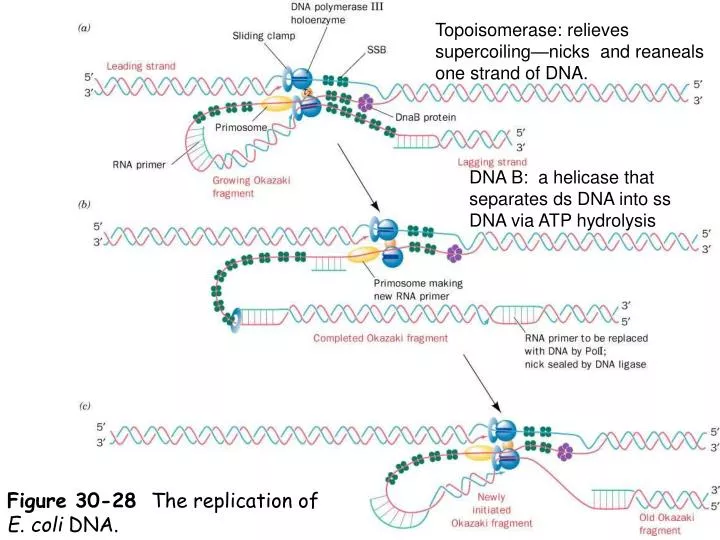
- DnaB (Helicase) Rekrutierung: Nach der Bindung von DnaA wird die DnaB-Helicase rekrutiert. DnaB ist ein Enzym, das die Wasserstoffbrücken zwischen den Basenpaaren aufbricht und die DNA-Doppelhelix auftrennt. Um die Helicase an die DNA zu binden, ist das DnaC-Protein erforderlich, welches als Helicase-Loader fungiert.
- Einzelstrang-Bindeproteine (SSB): Sobald die DNA getrennt ist, binden Einzelstrang-Bindeproteine (SSB) an die einzelnen DNA-Stränge, um zu verhindern, dass sie sich wieder miteinander verbinden. SSB stabilisieren die getrennten Stränge und halten sie für die Replikationsmaschinerie zugänglich.
Elongation der DNA
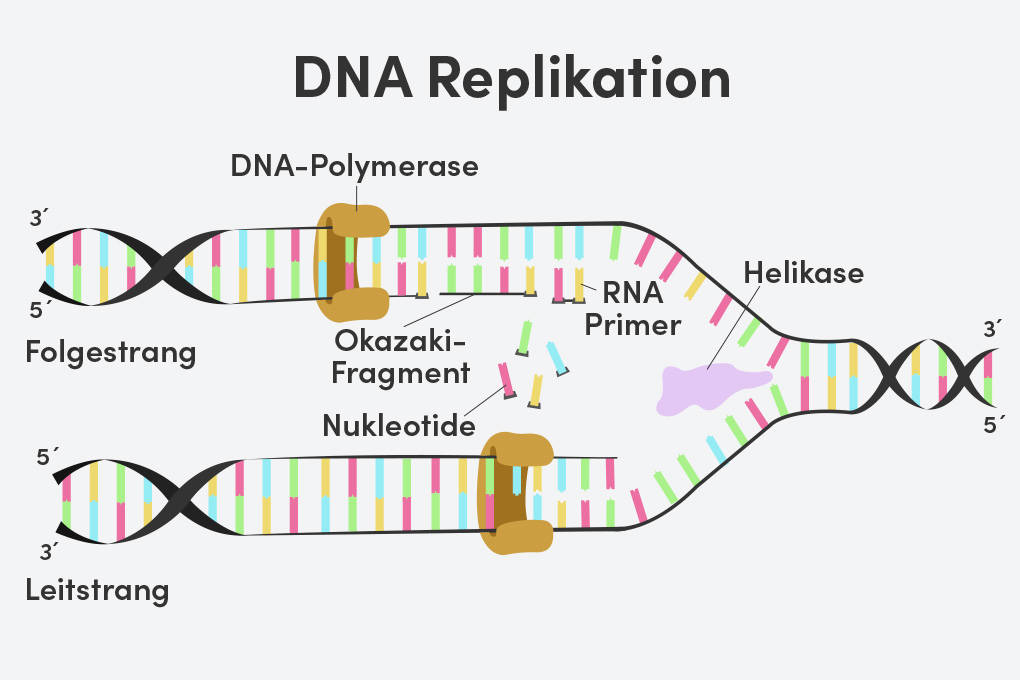
Nach der Initiation beginnt die Elongation, der eigentliche Prozess der DNA-Synthese. Dieses Stadium wird von dem Enzym DNA-Polymerase katalysiert. In E. coli gibt es verschiedene Arten von DNA-Polymerasen, aber die wichtigste für die Replikation ist die DNA-Polymerase III.
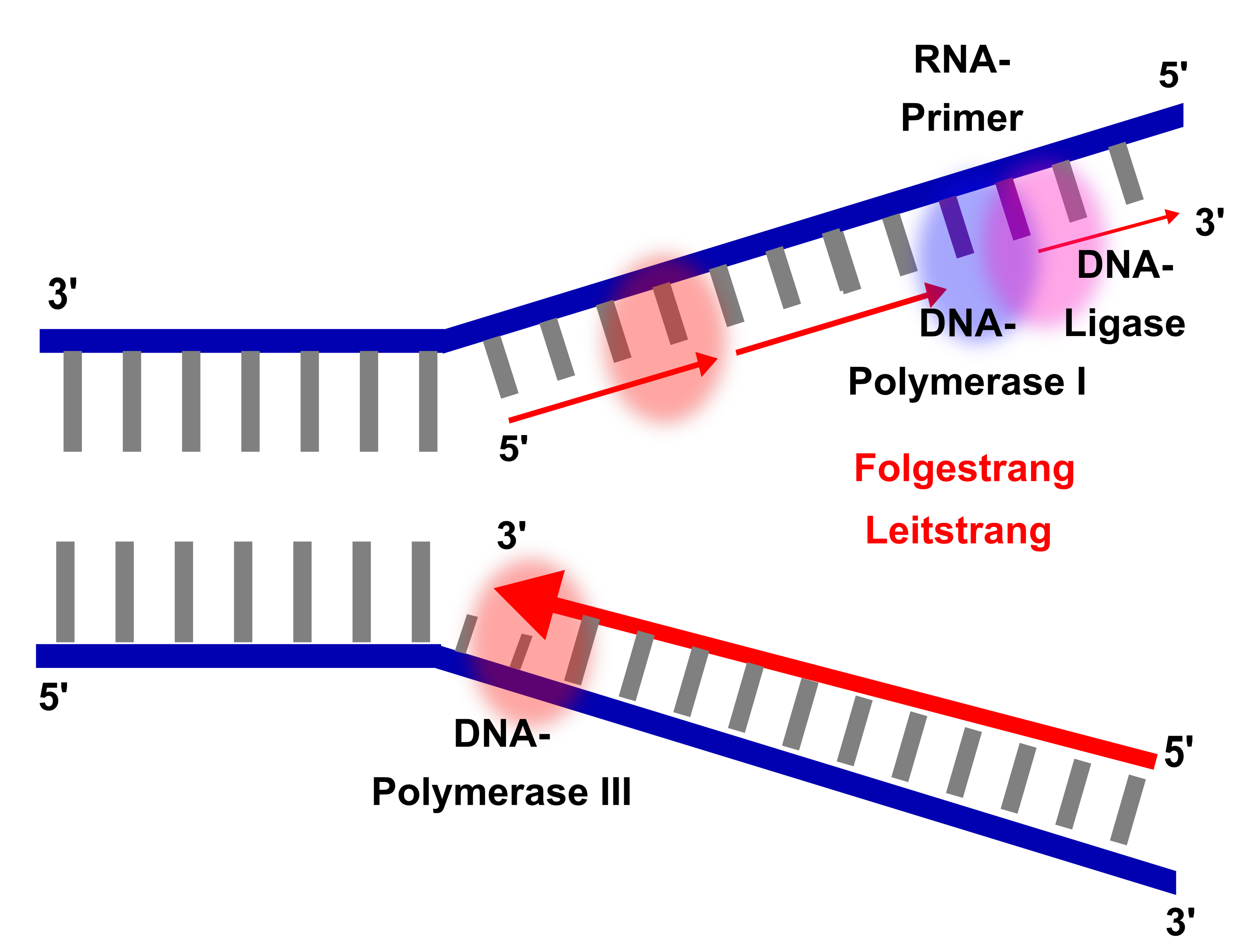
Die DNA-Polymerase III kann DNA nur in der 5'-zu-3'-Richtung synthetisieren, d.h. sie fügt Nukleotide nur an das 3'-Ende eines bestehenden Strangs hinzu. Dies führt zu einem wichtigen Unterschied in der Art und Weise, wie die beiden DNA-Stränge repliziert werden:
- Leitstrang (Leading Strand): Der Leitstrang wird kontinuierlich in Richtung der Replikationsgabel synthetisiert. Die DNA-Polymerase III kann einfach an den 3'-OH-Ende eines Primers (einer kurzen RNA-Sequenz) binden und die DNA kontinuierlich verlängern.
- Verzögerungsstrang (Lagging Strand): Der Verzögerungsstrang wird diskontinuierlich in entgegengesetzter Richtung der Replikationsgabel synthetisiert. Da die DNA-Polymerase III nur in der 5'-zu-3'-Richtung synthetisieren kann, wird der Verzögerungsstrang in kurzen Fragmenten synthetisiert, die als Okazaki-Fragmente bezeichnet werden. Für jedes Okazaki-Fragment ist ein neuer RNA-Primer erforderlich.
Der Prozess der Elongation am Verzögerungsstrang ist komplexer und erfordert die Koordination mehrerer Enzyme:
- Primase (DnaG): Die Primase ist eine RNA-Polymerase, die kurze RNA-Primer auf dem Verzögerungsstrang synthetisiert. Diese Primer dienen als Startpunkte für die DNA-Polymerase III.
- DNA-Polymerase III: Die DNA-Polymerase III verlängert die Okazaki-Fragmente, indem sie Nukleotide an die 3'-Ende der Primer hinzufügt.
- DNA-Polymerase I: Die DNA-Polymerase I hat eine 5'-zu-3'-Exonukleaseaktivität, d.h. sie kann RNA-Primer entfernen und durch DNA-Nukleotide ersetzen.
- DNA-Ligase: Die DNA-Ligase katalysiert die Bildung einer Phosphodiesterbindung zwischen dem 3'-OH-Ende eines Okazaki-Fragments und dem 5'-Phosphatende des nächsten Fragments und schließt so die Lücken zwischen den Okazaki-Fragmenten.
Die Rolle des Replisoms
Die effiziente und koordinierte Replikation erfordert eine komplexe molekulare Maschinerie, die als Replisom bezeichnet wird. Das Replisom besteht aus der DNA-Polymerase III, der Helicase (DnaB), der Primase (DnaG), SSB und anderen Proteinen. Das Replisom ermöglicht eine simultane Synthese von Leit- und Folgestrang. Dies wird durch die Schleifenbildung des Folgestrangs erreicht, wodurch beide Polymerasen (für Leit- und Folgestrang) in räumlicher Nähe bleiben. Diese räumliche Nähe erhöht die Geschwindigkeit und Effizienz der Replikation.
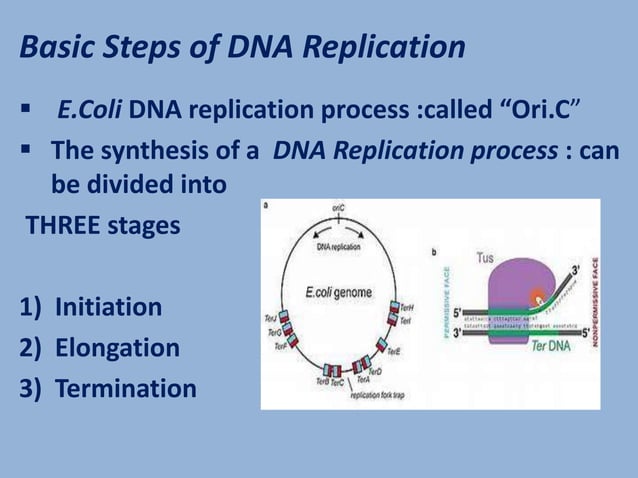
Termination der Replikation
Die DNA-Replikation in E. coli endet, wenn sich die beiden Replikationsgabeln am Terminationsbereich des Chromosoms treffen. Dieser Bereich enthält mehrere ter-Sequenzen, die von Tus-Proteinen (Termination Utilization Substance) gebunden werden. Die Tus-Proteine wirken als "Replikationsstoppschilder" und blockieren die weitere Bewegung der Replikationsgabeln.
Sobald die Replikation abgeschlossen ist, liegen zwei identische DNA-Moleküle vor, die miteinander verknäuelt sein können. Das Enzym Topoisomerase IV löst diese Verknäuelung, indem es vorübergehend DNA-Stränge durchtrennt und wieder miteinander verbindet. Dies ermöglicht die Trennung der beiden Tochterchromosomen und ihre Verteilung auf die Tochterzellen während der Zellteilung.
Genauigkeit und Fehlerkorrektur
Die DNA-Replikation ist ein hochgenauer Prozess, aber Fehler können dennoch auftreten. Die DNA-Polymerase III hat eine eingebaute Korrekturlesefunktion. Das bedeutet, dass sie falsche Nukleotide während der Synthese erkennen und entfernen kann. Diese Korrekturlesefunktion reduziert die Fehlerrate erheblich.
Neben der Korrekturlesefunktion der DNA-Polymerase gibt es auch andere DNA-Reparaturmechanismen, die dazu beitragen, Fehler, die während der Replikation auftreten können, zu korrigieren. Diese Reparaturmechanismen gewährleisten die Stabilität des Genoms von E. coli.
Zusammenfassung
Die DNA-Replikation in Escherichia coli ist ein komplexer und hochregulierter Prozess, der für die genetische Kontinuität unerlässlich ist. Er beginnt an einem spezifischen Replikationsursprung (oriC), erfordert die Beteiligung zahlreicher Enzyme wie DnaA, DnaB (Helicase), DNA-Polymerase III, Primase und DNA-Ligase. Der Prozess ist semikonservativ, wobei jede Tochter-DNA-Doppelhelix einen elterlichen und einen neu synthetisierten Strang enthält. Die Replikation verläuft bidirektional von oriC aus, wobei ein Leitstrang kontinuierlich und ein Verzögerungsstrang diskontinuierlich in Okazaki-Fragmenten synthetisiert wird. Die Termination erfolgt, wenn sich die Replikationsgabeln an den ter-Sequenzen treffen und die verknäuelten Chromosomen durch Topoisomerase IV getrennt werden. Die DNA-Polymerase III verfügt über eine Korrekturlesefunktion, und zusätzliche DNA-Reparaturmechanismen tragen zur Aufrechterhaltung der genetischen Genauigkeit bei. Das Verständnis dieses Prozesses ist entscheidend für die Biologie von Bakterien und bietet Einblicke in grundlegende molekulare Mechanismen.